반응형
분산분석이란
분산분석은 세 집단 이상의 모평균 차이를 검정하고, 표본에서의 차이가 통계적으로 유의한지 검증하기 위해 분산을 이용하는 방법을 말한다. 평균을 비교하는데 분산을 사용하는 이유는, 분산이 클 수록 치우쳐진 정도가 커 평균이 다를 확률이 높아지기 때문이다. 보통 분산분석은 "같다" 또는 "같지 않다"만 파악할 뿐, 다른 가설 검정처럼 "크다", "작다"는 파악하지 않는다. 여러 실험이나 연구에서 집단이 3개 이상인 경우가 많아 분산분석은 나름 활용도가 높다.
분산분석은 각 집단을 나누는 요인이 1개이면 일원분산분석(One-way ANOVA),

분산분석의 가설 설정
일반적인 가설검정에서 "같지 않다"는 방향을 모른다는 표현으로 양측검정을 하지만, 분산분석에서는 "평균이 다르다"는 의미로 쓰여 양측검정을 하지 않는다. 또한 우측검정이 좌측검정보다 사용하기가 편해서 F통계량이 작게 나와 좌측검정을 해야 할 경우에는 그냥 집단의 순서를 바꿔준다.
분산분석의 가정
- 독립성
- 각 관찰값이 독립적이어야 하는데, 일반적으로 수집한 관찰값들은 독립적으로 수집되었다고 보기 때문에 독립성 검정은 따로 진행하지 않는 편이다.
- 독립성 위배 시에는 반복측정 분산분석을 사용한다.
- 정규성
- Shapiro(소규모 샘플), Anderson-Darling(대규모 샘풀), Kolmogorov-Smironov, Jarque-Bera, QQ plot 등으로 검정한다.
- 히스토그램, 박스플롯, QQ플롯 등 시각화 자료로 많이 하는 편이라고 한다.
- 또한 정규분포라고 보기 곤란한 경우 로그 변환을 사용하기도 한다.
# 정규성 검정
from scipy.stats import norm
from scipy.stats import shapiro, anderson, jarque_bera, kstest, zscore, probplot
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
data = norm.rvs(size=100, random_state=0) #정규성 만족 샘플
data1 = np.random.rand(100) # 정규성을 만족할지 모르는 샘플(난수)
# shapiro 검정, 검정통계량과 P-value를 반환
sstat, sp = shapiro(data)
sstat1, sp1 = shapiro(data1)
# darling anderson, 검정통계량, 임계치, 유의수준 반환
dstat, cv, sl = anderson(data, dist='norm')
dstat1, cv1, sl1 = anderson(data, dist='norm')
#kstest(Kolmogorov-Smironov), 검정통계량, p-value를 반환
kstat, kp = kstest(data, cdf = norm.cdf)
kstat1, kp1 = kstest(data1, cdf = norm.cdf)
# jarque_bera, 검정통계량과 p-value, 왜도, 첨도를 반환
jstat, jp = jarque_bera(data)
jstat1, jp1 = jarque_bera(data1)
print('정규성 만족 샘플 정규성 검정 결과')
print(f'Shapiro : {sstat:.3f} (p : {sp:.3f})')
print(f'Anderson : {dstat:.3f} (critical value {cv[2]:.3f} significant level {sl[2]*0.01:.2f})')
print(f'Kstest_noraml : {sstat:.3f} (p {kp:.3f})')
print('\n')
print('무작위 샘플 정규성 검정 결과')
print(f'Shapiro : {sstat1:.3f} (p : {sp1:.3f})')
print(f'Anderson : {dstat1:.3f} (critical value {cv1[2]:.3f} significant level {sl1[2]*0.01:.2f})')
print(f'Kstest_noraml : {sstat1:.3f} (p {kp1:.3f})')
# QQ Plot: 데이터들이 대각선에 붙어 있을 수록 정규성을 만족하는 것으로 본다.
zdata = zscore(data)
fig, ax = plt.subplots(2, 2, figsize=(20, 10))
(osm, odr), (slope, intercept, r) = probplot(zdata, plot=ax[0, 0])
ax[0, 0].set_title('Norm sample QQ plot', fontsize = 20)
ax[0, 1].hist(data)
ax[0, 1].set_title('Norm sample histogram', fontsize = 20)
zdata1 = zscore(data1)
(osm1, odr1), (slope1, intercept1, r1) = probplot(zdata1, plot=ax[1, 0])
ax[1, 0].set_title('Norm sample QQ plot', fontsize = 20)
ax[1, 1].hist(data1)
ax[1, 1].set_title('Norm sample histogram', fontsize = 20)
plt.show()
- 등분산성
- 정규성을 만족할 경우 Bartlett, 만족하지 않을 경우 Levene검정.
- 모든 데이터는 평균이 달라도 분산이 동일한 모집단이라는 가정이다.
- 가장 큰 분산과 가장 작은 분산의 비가 4:1이 넘지 않으면 사용해도 된다고 한다.
- 등분산성을 만족하지 않는 경우 Welch ANOVA를 적용한다.
# 정규성을 만족한 Bartlett 검정
from scipy.stats import norm
from scipy.stats import bartlett
data1 = norm.rvs(size=100, random_state=0)
data2 = norm.rvs(size=100, random_state=1)
data3 = norm.rvs(size=100, random_state=2)
bstat, p = bartlett(data1, data2, data3)
print('라이브러리 검정')
print(f'stat : {bstat}, p : {p}')
# "분산이 모두 같다"가 귀무가설이므로 p-value가 유의수준보다 크면 귀무가설을 기각하지 않고 등분산으로 가정됌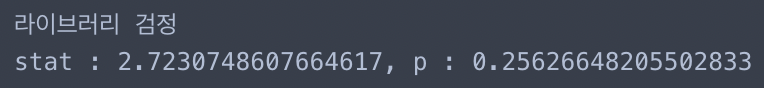
반응형
'Statistics' 카테고리의 다른 글
| 일원분산분석 (One-way ANOVA)(feat. Python) (0) | 2024.01.23 |
|---|---|
| 분산분석(ANOVA) (0) | 2024.01.23 |
| 이표본 검정(Two-sample)(feat.Python) (1) | 2024.01.17 |
| 일표본(One-sample) 검정(feat.Python) (0) | 2024.01.16 |
| 추정과 가설 검정 간단 정리 (0) | 2024.01.15 |



